No curso de Sistemas de Informação, não temos uma disciplina específica para estudar Bioinformática. Porém, para aqueles que gostam do tema e que tem a intenção de estudar essa área em algum outro momento (numa Iniciação Científica ou em algum curso extracurricular, por exemplo), é preciso estar ciente de que muitos dos conceitos básicos necessários para o entendimento da área são estudados nas disciplinas do nosso curso. Assim, elaboramos esta matéria como uma forma de adiantar quais são estes conceitos (para que você fique esperto quando os estiver estudando nas matérias regulares do nosso curso).
Vivian Mayumi Yamassaki Pereira
Algumas vezes durante a graduação, não conseguimos perceber o porquê de estudarmos certos tópicos em algumas disciplinas. Às vezes, só percebemos sua aplicabilidade quando iniciamos uma iniciação científica, mestrado/doutorado ou quando começamos a trabalhar na indústria de software e nas organizações. Também podemos encontrá-la quando assistimos palestras ou participamos de cursos de verão.
Um curso de verão é um curso dado no período de férias que, no caso da USP, ocorre entre os meses de dezembro e fevereiro. É uma oportunidade para que o aluno se atualize ou mesmo complemente sua formação com um conteúdo que não está presente na grade curricular de seu curso de graduação. Há diversos cursos de verão oferecidos pela USP, como os oferecidos pelo IME [1], por exemplo. Alguns também são oferecidos pela EACH e por Programas de Pós-graduação, por isso, é interessante monitorar os respectivos websites para verificar o seu oferecimento.
Como exemplo de um curso de verão, podemos citar o Curso de Verão de Bioinformática do Programa Interunidades de Pós-Graduação de Bioinformática da USP, ocorrido no início deste ano. Este curso de verão teve como objetivo “contribuir para a introdução dos participantes na área de Bioinformática, abordando os principais temas da área. O objetivo, em particular, é proporcionar uma preparação básica aos potenciais candidatos ao programa de mestrado e/ou doutorado em bioinformática, oferecido pelo programa Interunidades da USP” [2]. Duas integrantes do PET-SI¹ participaram do curso e durante o mesmo puderam perceber o uso de vários conceitos que estudamos previamente em diferentes disciplinas do nosso curso na área de Bioinformática.
Nesta matéria, alguns dos exemplos dados no Curso de Verão serão utilizados para demonstrar que aquilo que nós estudamos em SI pode ser aplicado na resolução de inúmeros problemas.
Uma das disciplinas em que muitos conceitos necessários para resolução de problemas em Bioinformática são apresentados é a de Algoritmos e Estruturas de Dados II (AED II, ministrada no terceiro semestre do curso). Citando um exemplo, vimos que a Teoria dos Grafos pode ser utilizada para mapear interações que ocorrem no cérebro e, com isso, verificar quais áreas estão envolvidas em uma determinada doença.
Além disso, vale mencionar que hoje em dia, existem diversos bancos de dados voltados para a Bioinformática, como por exemplo, o GenBank [3]. Este banco de dados armazena sequências genéticas do Centro Nacional de Informação Biotecnológica dos Estados Unidos. Assim como ele, outros bancos de dados dessa área devem ser muito bem modelados. Isso porque com os avanços da tecnologia, tornou-se mais rápido e barato realizar o sequenciamento de DNAs e, com isso, houve um aumento exponencial do volume de dados gerados. Por este motivo, para quem quer trabalhar com sequenciamento e interpretação de DNAs, é muito importante atentar para a disciplina de Banco de Dados (BD, ministrada no quarto semestre), que fornece conceitos essenciais para a área, tais como o conhecimento para: escolher qual a melhor maneira de armazenar dados, como as sequências gigantescas do DNA de um organismo; modelar o banco de dados de maneira a atender às suas necessidades; e otimizar os procedimentos de buscas de modo a melhorar a performance da recuperação da informação.
Já em relação à Inteligência Artificial (IA, ministrada no quinto semestre), vimos que conceitos de Reconhecimento de Padrões trabalhados na disciplina embasam o entendimento dos procedimentos que executam estimações de expressão gênica. Há diversas técnicas de Inteligência Artificial que são utilizadas para realizar a análise de microarray, amplamente utilizada em Bioinformática, tais como algoritmos genéticos e redes neurais artificiais [4].
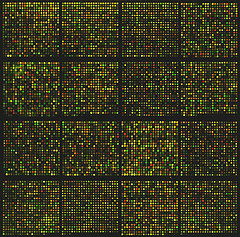
Com a análise de microarray², é possível analisar a diferença de expressão gênica de células normais e células cancerígenas, por exemplo. Para implementar o procedimento geral para realizar essa análise, utiliza-se o cDNA (DNA complementar) extraído das amostras dessas células, que são rotulados com corantes fluorescentes diferentes para que seja possível distinguir os níveis de expressão gênica de cada amostra. Os cDNAs das amostras são então hibridizados com vários genes, conhecidos, presentes em um array disposto em uma lâmina de vidro. Depois, este array é escaneado e é gerada uma imagem colorida como a da Figura 1. Ao analisar padrões de expressão gênica presentes nesta imagem, é possível verificar quais genes estão envolvidas nesse tumor. Supondo que as células cancerígenas tenham sido rotuladas com o corante vermelho, pode-se concluir que, nos locais do array que estão apenas vermelhos, houve um significante nível de expressão gênica nas células cancerígenas, enquanto que nas células normais, não.
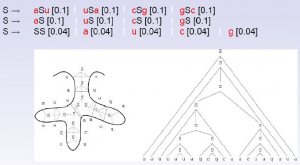
Também vimos as aplicações de conceitos ministrados em Introdução à Teoria da Computação (ITC, ministrada no quarto semestre): as Gramáticas Livres de Contexto Estocásticas, usadas para fazer a predição de estruturas secundárias de RNAs, por exemplo. Tal exemplo de sua utilização pode ser visualizado na Figura 2. Nela, é possível observar as regras da gramática, cujo alfabeto é formado pelas letras que representam as bases nitrogenadas que compõem um RNA (a – adenina, c – citosina, g – guanina e u – uracila), e suas probabilidades de ocorrência. Uma das possíveis árvores de derivação dessa gramática encontra-se à direita e, ao lado esquerdo, podemos observar essa derivação na forma de uma estrutura secundária.
Por fim, há ainda a utilização da Programação Dinâmica para encontrar alinhamentos ótimos entre duas sequências de DNA, para verificar o quão parecidas elas são e, com isso, entender a história evolutiva de genes e organismos. Apesar de não estudarmos especificamente sobre Programação Dinâmica em SI, conceitos aprendidos em Introdução à Análise de Algoritmos (IAA, ministrada no segundo semestre) são a base para aprendê-la.
Claro que além da área de Bioinformática, o que vimos nas disciplinas do curso de Sistemas de Informação têm importância em inúmeras outras áreas do conhecimento. Por isso, é interessante procurar conhecer as aplicabilidades daquilo que nós estudamos, tanto para adquirir mais conhecimento, quanto para incentivar os estudos. Se você tem dúvidas sobre a aplicabilidade de algumas matérias, peça a seu professor algumas bibliografias onde você pode estudar mais a fundo cada quesito. Ou, se você achar interessante, você pode nos ajudar a escrever uma matéria falando da aplicabilidade dos conceitos de uma determinada disciplina. Podemos publicar essa nova matéria no site do nosso jornal.
Notas
¹ As petianas Vivian e Camila participaram do curso para adquirir alguns conhecimentos úteis para o desenvolvimento de seus trabalhos de Iniciação Científica.
As referidas bolsistas agradecem o apoio da Pró-Reitoria de Graduação, no financiamento de algumas atividades realizadas pelo grupo PET-SI, incluindo a participação neste curso de verão.
² Caso queira saber um pouco mais sobre a técnica de análise de microarray, não deixe de acessar este simulador desenvolvido pela Universidade de Utah, que simula e explica, com mais detalhes, como a técnica funciona: http://learn.genetics.utah.edu/content/labs/microarray.
Referências
[1] IME- USP : Programa de Verão. Disponível em <http://www.ime.usp.br/verao>.
[2]Curso de Verão 2014 – Bioinformática. Disponível em <http://www.ime.usp.br/posbioinfo/cv2014/>.
[3] GenBank. Disponível em <https://www.ncbi.nlm.nih.gov/genbank/>.
[4] VALAFAR, F. 2002. Pattern Recognition Techniques in Microarray Data Analysis: A Survey. Special Issue Annals of New York Academy of Sciences. Techniques in Bioinformatics and Medical Informatics. Volume 980, 41-64.